learn_pul
A Minimal Example of PU Learning Using R
李家翔 2019-02-26
数据来自
from sklearn.datasets.samples_generator import make_blobs
以下实验的目的是检验当部分正样本和全部负样本未知的情况下,比较PU Learning 的模型效果和 已经全部正负样本下的模型结果。
如果两个结果差异不大,表示 PU Learning 明显效果不错。
suppressMessages(library(tidyverse))
x <- read_csv("datasets/make_circles_x.csv")
## Parsed with column specification:
## cols(
## feature1 = col_double(),
## feature2 = col_double()
## )
y <- read_csv("datasets/make_circles_y.csv")
## Parsed with column specification:
## cols(
## y = col_double()
## )
table(y)
## y
## 0 1
## 3000 3000
head(x)
## # A tibble: 6 x 2
## feature1 feature2
## <dbl> <dbl>
## 1 0.789 3.79
## 2 6.66 5.88
## 3 0.440 -1.47
## 4 0.334 4.32
## 5 4.92 4.28
## 6 5.82 2.74
假设 2700 个正样本是未知的,所有的负样本是未知的。
set.seed(123)
unlabelled_index <- sample(x = which(y == 1),size = 2700,replace = F)
y_new <- y
y_new[unlabelled_index,]$y <- 0
data <- bind_cols(y_new,x)
table(data$y)
##
## 0 1
## 5700 300
table(y)
## y
## 0 1
## 3000 3000
library(randomForest)
## randomForest 4.6-14
## Type rfNews() to see new features/changes/bug fixes.
##
## Attaching package: 'randomForest'
## The following object is masked from 'package:dplyr':
##
## combine
## The following object is masked from 'package:ggplot2':
##
## margin
model <- randomForest(as.factor(y) ~ feature1 + feature2, data = data)
yhat <- predict(model,type = 'prob')[,2]
ks_table <- function(yhat, y) {
library(tidyverse)
pred <- ROCR::prediction(yhat, y)
perf <- ROCR::performance(pred, 'tpr', 'fpr')
# tpr true positive rate
# fpr false positive rate
perf_df <- data.frame(perf@x.values, perf@y.values)
names(perf_df) <- c("fpr", "tpr")
per_df2 <-
perf_df %>%
mutate(cutoff = 1:nrow(.)/nrow(.)) %>%
mutate(ks = tpr - fpr)
return(per_df2)
}
ks_table(yhat,y) %>%
gather(key,value, tpr,fpr,ks) %>%
ggplot(aes(x = cutoff, y = value, col = key)) +
geom_line()
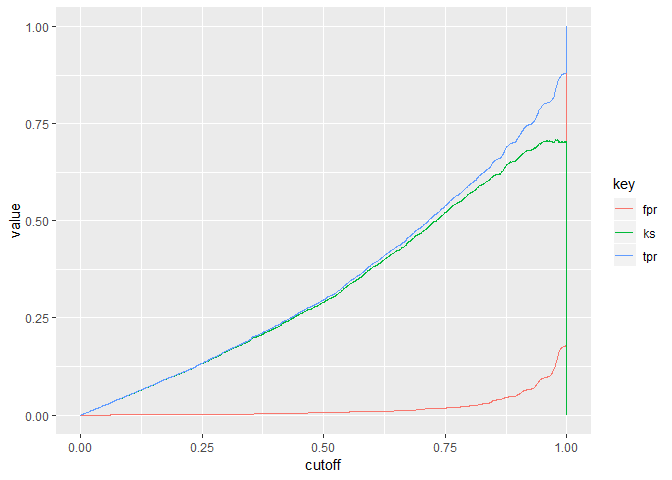
看到 KS 可以达到 70% 左右,因此模型效果并不差。
那么假设正样本和负样本完全已知的情况下,对比简单模型效果。
data2 <- bind_cols(y,x)
model2 <- randomForest(as.factor(y) ~ feature1 + feature2, data = data2)
yhat2 <- predict(model2,type = 'prob')[,2]
ks_table(yhat2,y) %>%
gather(key,value, tpr,fpr,ks) %>%
ggplot(aes(x = cutoff, y = value, col = key)) +
geom_line()
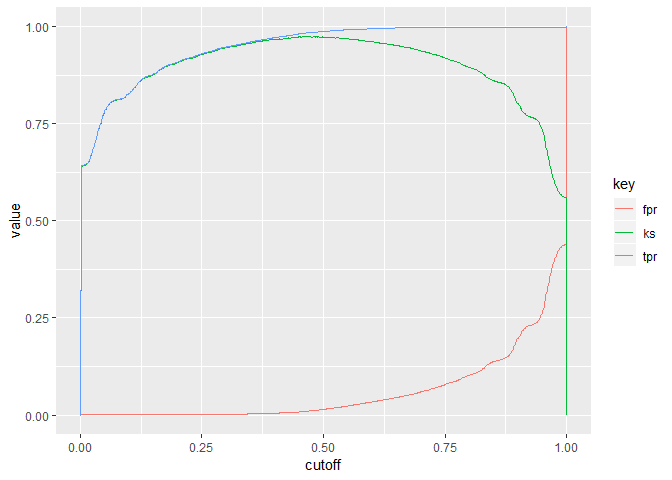
KS 为 100%,这是因为 feature1 和 feature2 本身是完全有效的两个变量。 对比前者 KS 差异 1/3 的准确性,因此是可以接受的。
PU Learning 的结果并不差。